预约演示
更新于:2025-08-29

TransGen Biotech Co., Ltd.
更新于:2025-08-29
概览
关联
1
项与 北京全式金生物技术股份有限公司 相关的临床试验NCT05492227
SARS-CoV-2 Nucleic Acid Detection Kit
SARS-CoV-2 Nucleic Acid Detection Kit is an In Vitro Diagnostic (IVD) medical device intended for RNA detection of SARS-CoV-2 in human specimens using multiplex real-time polymerase chain reaction (RT-PCR) technology. The conserved regions of ORF1ab and N genes are used as target sites of the primers and probes according to the indications provided in the Instructions for Use (IFU).
开始日期2022-07-05 |
申办/合作机构 |
100 项与 北京全式金生物技术股份有限公司 相关的临床结果
登录后查看更多信息
0 项与 北京全式金生物技术股份有限公司 相关的专利(医药)
登录后查看更多信息
1
项与 北京全式金生物技术股份有限公司 相关的文献(医药)2021-08-31·Proceedings of the National Academy of Sciences of the United States of America1区 · 综合性期刊
ChlamydomonasLZTFL1 mediates phototaxis via controlling BBSome recruitment to the basal body and its reassembly at the ciliary tip
1区 · 综合性期刊
Article
作者: Sun, Wei-Yue ; Zhang, Rui-Kai ; Liu, Yan-Xia ; Xin, Wen ; Li, Rong-Chao ; Xue, Bin ; Wu, Mingfu ; Fan, Zhen-Chuan
Many G protein-coupled receptors and other signaling proteins localize to the ciliary membrane for regulating diverse cellular processes. The BBSome composed of multiple Bardet-Biedl syndrome (BBS) proteins is an intraflagellar transport (IFT) cargo adaptor essential for sorting signaling proteins in and/or out of cilia via IFT. Leucine zipper transcription factor-like 1 (LZTFL1) protein mediates ciliary signaling by controlling BBSome ciliary content, reflecting how LZTFL1 mutations could cause BBS. However, the mechanistic mechanism underlying this process remains elusive thus far. Here, we show that LZTFL1 maintains BBSome ciliary dynamics by finely controlling BBSome recruitment to the basal body and its reassembly at the ciliary tip simultaneously in Chlamydomonas reinhardtii LZTFL1 directs BBSome recruitment to the basal body via promoting basal body targeting of Arf-like 6 GTPase BBS3, thus deciding the BBSome amount available for loading onto anterograde IFT trains for entering cilia. Meanwhile, LZTFL1 stabilizes the IFT25/27 component of the IFT-B1 subcomplex in the cell body so as to control its presence and amount at the basal body for entering cilia. Since IFT25/27 promotes BBSome reassembly at the ciliary tip for loading onto retrograde IFT trains, LZTFL1 thus also directs BBSome removal out of cilia. Therefore, LZTFL1 dysfunction deprives the BBSome of ciliary presence and generates Chlamydomonas cells defective in phototaxis. In summary, our data propose that LZTFL1 maintains BBSome dynamics in cilia by such a dual-mode system, providing insights into how LZTFL1 mediates ciliary signaling through maintaining BBSome ciliary dynamics and the pathogenetic mechanism of the BBS disorder as well.
38
项与 北京全式金生物技术股份有限公司 相关的新闻(医药)2025-08-08
文章信息
文章题目
Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales
期刊
Cell
发表时间
2025 年 8 月 4 日
主要内容
中国科学院遗传与发育生物学研究所高彩霞团队在 Cell 杂志上在线发表了题为“Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales”的研究论文,系统报道了一种新型可编程的染色体水平大片段 DNA 精准操纵技术 PCE 。该技术在动植物中实现了从千碱基到兆碱基级别 DNA 的多种类型且精准无痕的编辑,显著提升了真核生物基因组的操纵尺度和能力。
原文链接
https://doi-org.libproxy1.nus.edu.sg/10.1016/j.cell.2025.07.011
使用 TransGen 产品
TransDetect® PCR Mycoplasma Detection Kit (FM311)
Ampicillin (GG101)
Kanamycin (GG201)
研究背景
基因组编辑技术作为生命科学领域的一项革命性突破,为基础研究和应用开发提供了强大的技术支撑。以 CRISPR 及其衍生技术为代表的基因编辑平台,通过向导 RNA 精确引导核酸酶靶向基因组特定位点,已广泛应用于特定碱基和短片段 DNA 的精准编辑。然而,大片段 DNA 编辑仍存在显著技术瓶颈,尤其是数千至数百万碱基规模的精准操纵仍是领域内亟待解决的核心难题。攻克这一技术壁垒将为基因组大尺度操作提供关键支持——包括基因关键区域的替换与修复、功能基因序列的完整插入、基因簇的删减或重定位,以及人工构建基因组结构变异等。这一突破不仅能推动遗传操作技术的革新,还将为疾病治疗和作物改良领域带来重大进展。理想的大片段 DNA 编辑工具应能实现染色体水平的多类型编辑,涵盖精准插入、替换、删除、倒位和易位等操作。但现有工具在编辑效率、操作尺度、精准度及功能多样性等方面仍存在明显不足。
文章概述
Cre-Lox 重组系统虽然具备染色体水平 DNA 编辑能力,但在实际应用中受到三个关键问题的制约:首先,Lox 位点的对称结构导致重组反应可逆,显著降低了目标编辑效率;其次,Cre 酶需要形成四聚体才能发挥作用,提升其活性的工程改造难度高;最后,重组后特异性位点残留,影响编辑产物的精确性。
研究团队针对现有技术瓶颈制定了分步突破策略。首先,开发了高通量重组位点快速改造平台,并提出不对称 Lox 位点设计原则,成功创制新型 Lox 变体,其在保持高效正向重组能力的同时,将可逆重组活性显著降低至接近阴性对照水平。其次,进一步利用自主研发的融合蛋白通用逆折叠模型、结构与进化约束信息的蛋白定向进化平台 AiCE,对 Cre 蛋白的多聚化界面进行精确改造,成功获得了重组活性提高 3.5 倍的工程化 Cre 蛋白变体。最后,创建并优化了重组酶的无痕编辑策略 Re-pegRNA,利用引导编辑器的高效编辑特性,通过设计特异性 pegRNA 对重组后残留的 Lox 位点进行“重引导编辑”(re-prime editing),将其精准替换为原有基因组序列。
基于这些技术突破,研究团队成功开发了 PCE 和 RePCE 两大可编程染色体编辑系统,可对不同 Lox 位点的插入位置和方向进行灵活编程,实现从 kb 到 Mb 尺度的大片段 DNA 精准无痕操纵。实验验证表明,该系统在动植物细胞中可高效完成多种基因组操作,包括 18.8 kb 大片段 DNA 的定点整合、5 kb 序列的定向替换、12 Mb 的染色体倒位、4 Mb 的染色体删除及整条染色体的易位,并利用该技术创制了含 315 kb 精准倒位的抗除草剂水稻种质。
综上,该研究开发了新型染色体编辑系统 PCE,在动植物中实现了跨越 kb 到 Mb 级别的多类型染色体精准操纵。该工具不仅能实现多基因叠加,还可通过操控基因组结构变异,为作物性状改良和遗传疾病治疗开辟新路径。此外,该技术有望推动新型育种策略的发展,例如通过操纵遗传连锁、调控重组频率实现育性控制,以及消除连锁累赘,充分释放野生种质资源中优异等位基因的育种潜力。精准染色体编辑技术的突破将加速人工染色体构建,在合成生物学等新兴领域也有重要的应用前景。
PCE 系统的开发和精准染色体编辑概述
全式金生物产品支撑
优质的试剂是科学研究的利器。全式金生物的支原体检测试剂盒(PCR法)(FM311)、氨苄青霉素(GG101)、卡那霉素(GG201)助力本研究。产品自上市以来,深受客户青睐,多次荣登知名期刊,助力科学研究。
TransDetect® PCR Mycoplasma Detection Kit (FM311)
一款通过 PCR 方法检测培养细胞等生物材料中的支原体,针对支原体 16S rRNA 序列保守区域设计特异引物,直接使用细胞培养液作为模板,特异性扩增支原体 DNA。
产品特点
★ 检测灵敏度高、准确性好。
★ 特异性强,只扩增支原体 DNA。
★ 操作简便,无需提取基因组 DNA。
Ampicillin (GG101)
Ampicillin 为青霉素的衍生物,可通过干扰细菌的细胞壁合成来杀死生长细胞。其抗性机制为抗性基因 (Bla) 特异性表达外周质酶—β-内酰胺酶,该酶可切割抗生素的 β-内酰胺环。
Kanamycin (GG201)
卡那霉素 (kanamycin) 为一种杀菌剂,通过结合于 70S 核糖体,引起 mRNA 的错读。其抗性机制为抗性基因 (Kan) 特异性表达氨基糖苷磷酸转移酶,可修饰抗生素,抑制其与核糖体结合。
产品文献集锦
使用 TransDetect® PCR Mycoplasma Detection Kit (FM311) 产品发表的部分文章
• Sun C, Li H C, Liu Y J, et al. Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales[J]. Cell, 2025.(IF 45.6)
• Liu J L, Yan X Q, Wu H, et al. RNA codon expansion via programmable pseudo uridine editing and decoding[J]. Nature, 2025.(IF 50.5)
• Fei H Y, LI Y J,Liu Y J, et al. Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints[J]. Cell, 2025.(IF 45.6)
• Nian Z, Dou Y, Shen Y, et al. Interleukin-34-orchestrated tumor-associated macrophage reprogramming is required for tumor immune escape driven by p53 inactivation[J]. Immunity, 2024.(IF 25.5)
• Xu J, Liang Y, Li N, et al. Clathrin-associated carriers enable recycling through a kiss-and-run mechanism[J]. Nature Cell Biology, 2024.(IF 17.3)
• Wang J, An Z, Wu Z, et al. Spatial organization of PI3K-PI (3, 4, 5) P3-AKT signaling by focal adhesions[J]. Molecular Cell, 2024.(IF 14.5)
• Huang J, Lin Q, Fei H, et al. Discovery of deaminase functions by structure-based protein clustering[J]. Cell, 2023.(IF 65.00)
• Lei Z, Meng H, Liu L, et al. Mitochondrial base editor induces substantial nuclear off-target mutations[J]. Nature, 2022.(IF 69.50)
• Nian Z, Zheng X, Dou Y, et al. Rapamycin pretreatment rescues the bone marrow AML cell elimination capacity of CAR-T cells[J]. Clinical Cancer Research, 2021.(IF 11.4)
• Xu D, Zhao H, Jin M, et al. Modulating TRADD to restore cellular homeostasis and inhibit apoptosis[J]. Nature, 2020.(IF 50.5)
使用 Ampicillin (GG101) 产品发表的部分文章
• Sun C, Li H C, Liu Y J, et al. Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales[J]. Cell, 2025.(IF 45.6)
• Liu L, Zou L, Li K, et al. Template-independent genome editing in the Pcdh15av− 3j mouse, a model of human DFNB23 nonsyndromic deafness[J]. Cell Reports, 2022. (IF 9.995)
• Jin S, Fei H, Zhu Z, et al. Rationally designed APOBEC3B cytosine base editors with improved specificity[J]. Molecular cell, 2020.(IF 15.58)
使用 Kanamycin (GG201) 产品发表的部分文章
• Sun C, Li H C, Liu Y J, et al. Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales[J]. Cell, 2025.(IF 45.6)
• Gong T, Hu C Y, Zhang C Q, et al. Catalytic Mechanism of Gut Benzyl Ether Reductase for Efficient Bioconversion of Furofuran Lignans into Enterolignan Precursors[J]. Journal of Agricultural and Food Chemistry, 2025. (IF 6.2)
• Jin S, Fei H, Zhu Z, et al. Rationally designed APOBEC3B cytosine base editors with improved specificity[J]. Molecular cell, 2020.(IF 15.58)
全式金生物的产品再度亮相 Cell 期刊,不仅是对全式金生物产品卓越品质与雄厚实力的有力见证,更是生动展现了全式金生物长期秉持的“品质高于一切,精品服务客户”核心理念。一直以来,全式金生物凭借对品质的执着追求和对创新的不懈探索,其产品已成为众多科研工作者信赖的得力助手。展望未来,我们将持续推出更多优质产品,期望携手更多科研领域的杰出人才,共同攀登科学高峰,书写科研创新的辉煌篇章。
临床研究基因疗法
2025-07-31
文章信息文章题目Deciphering phenylalanine-derived salicylic acid biosynthesis in plants期刊Nature发表时间2025 年 7 月 23 日主要内容浙江大学潘荣辉团队联合范鹏祥团队在 Nature 期刊发表了题为“Deciphering phenylalanine-derived salicylic acid biosynthesis in plants”研究论文。该研究揭示了水稻从苯甲酰辅酶 A 到水杨酸的酯化-羟化-水解(BEBT-BBH-BSE)三步酶联反应模块,打通了水稻的水杨酸合成途径,并进一步证明了该模块在其他农作物中也是广泛保守的。原文链接https://www-nature-com.libproxy1.nus.edu.sg/articles/s41586-025-09280-9使用 TransGen 产品EasyScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AE311)研究背景水杨酸(Salicylic acid,SA)早在 4000 多年前,就被人们在草药中用于消炎、镇痛,18 世纪首次从柳树皮中分离出来,基于它制成的阿司匹林(乙酰水杨酸)是人类历史上使用最广泛的药物。作为一种植物激素,水杨酸在植物免疫防御中起关键作用,植物通过异分支酸合成酶(ICS)和苯丙氨酸解氨酶(PAL)两条途径合成水杨酸,其中 ICS 途径已在拟南芥等多种植物中得到全面研究,但 PAL 途径的机制仍不完全清楚。文章概述潘荣辉团队基于前期对 CNL 功能的研究基础,结合团队在细胞器代谢功能研究方面的长期经验,运用基因共表达与蛋白生化技术,首次揭示了过氧化物酶体中的苯甲酰辅酶 A:苄醇苯甲酰转移酶(BEBT)能够催化苯甲酰辅酶 A 生成苯甲酸苄酯。通过同位素标记技术,研究人员颠覆性地证实苯甲酸苄酯才是水杨酸生物合成途径的关键中间体,而非传统认知中的苯甲酸。这一突破性发现打破了该领域 30 多年来关于苯甲酸直接羟化形成水杨酸的主流认知。明确水杨酸合成路径的关键节点后,研究团队通过共表达分析成功鉴定了苯甲酸苄酯羟化酶(BBH),填补了该领域羟化酶鉴定的空白。体外酶活实验显示,BBH 能够催化苯甲酸苄酯生成水杨酸苄酯。该羟化酶定位于内质网,BBH 功能缺失的突变体中水杨酸含量几乎为零,而外源施加水杨酸苄酯可恢复 BBH 突变体的水杨酸合成缺陷。随后,研究人员发现定位于细胞质的水杨酸苄酯水解酶(BSE)能够将水杨酸苄酯分解为水杨酸。通过构建了 BSE 功能缺失突变体,发现该突变体中水杨酸含量大幅下降。通过同位素示踪实验,进一步证实了 BBH 和 BSE 在水杨酸合成中的功能。研究团队在阐明水稻水杨酸(SA)合成通路后,进一步证实该途径对水稻抗稻瘟病菌的重要性。利用 VIGS 技术构建棉花、番茄和小麦的相关突变体,发现病原菌侵染诱导的 SA 合成在这些突变体中均受到显著抑制,同时 SA 信号通路下游抗病相关基因的表达水平也明显降低,证明了这一途径在这些重要作物中的功能保守性。在更广泛的不同植物中,新发现的 BEBT-BBH-BSE 模块均显著响应病菌,而拟南芥所在的十字花科是例外。这些研究系统地揭示了 BEBT-BBH-BSE 级联反应模块在包括水稻在内等大部分农作物的水杨酸生物合成途径中的关键作用。该研究不仅阐明了长期悬而未决的植物苯丙氨酸起源的 SA 生物合成的直接反应机制,还证实了这一模块在多种植物中的保守性,为作物抗病育种提供了新的理论基础和潜在靶点。这项研究填补了植物关键防御激素生物合成领域的一项重大知识空白,为培育抗病作物的新策略奠定了基础。全式金生物产品支撑优质的试剂是科学研究的利器。全式金生物的反转录试剂(AE311)助力本研究。产品自上市以来,深受客户青睐,多次荣登知名期刊,助力科学研究。EasyScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AE311)本产品以 RNA 为模板,在同一反应体系中,合成第一链 cDNA 的同时去除 RNA 模板中残留的基因组 DNA。反应结束后,只需在 85℃ 加热 5 秒钟,即可同时失活 TranScript® RT/RI 与 gDNA Remover。 产品特点 ★ 在同一反应体系中,同时完成反转录与基因组 DNA 的去除,操作简便,降低污染机率。★ 产物用于 qPCR:反转录 15 分钟;产物用于 PCR:反转录 30 分钟。★ 反应结束后,同时热失活 RT/RI 与 gDNA Remover。与传统的用 DNase I 预处理 RNA 的方法相比,避免了处理后热失活 DNase I 对 RNA 的损伤。★ 合成片段≤8 kb。产品文献集锦使用 EasyScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AE311)产品发表的部分文章• Wang Y K, Song S Y, Zhang W X, et al. Deciphering phenylalanine-derived salicylic acid biosynthesis in plants [J]. Nature, 2025. (IF 48.5)• Yu Y, Li W, Liu Y, et al. A Zea genus-specific micropeptide controls kernel dehydration in maize[J]. Cell, 2024.(IF 45.5)• Zheng Q, Xing J, Li X, et al. PRDM16 suppresses ferroptosis to protect against sepsis-associated acute kidney injury by targeting the NRF2/GPX4 axis[J]. Redox Biology, 2024.(IF 10.7)• Shi Q, Xia Y, Xue N, et al. Modulation of starch synthesis in Arabidopsis via phytochrome B‐mediated light signal transduction[J]. Journal of Integrative Plant Biology, 2024.(IF 9.3)• Lin J L, Chen L X, Wu W K, et al. Single-cell RNA sequencing reveals a hierarchical transcriptional regulatory network of terpenoid biosynthesis in cotton secretory glandular cells[J]. Molecular plant, 2023.(IF 17.1)• Lin J L, Fang X, Li J X, et al. Dirigent gene editing of gossypol enantiomers for toxicity-depleted cotton seeds[J]. Nature Plants, 2023.(IF 15.8)• Song M, Yao H, Sun Z, et al. METTL3/YTHDC1-medicated m6A modification of circRNA3634 regulates the proliferation and differentiation of antler chondrocytes by miR-124486-5-MAPK1 axis[J]. Cellular & Molecular Biology Letters, 2023.(IF 9.2)• Li N, Duan Y, Ye Q, et al. The Arabidopsis eIF4E1 regulates NRT1. 1-mediated nitrate signaling at both translational and transcriptional levels[J]. The New Phytologist, 2023.(IF 8.3)• Sheng C, Zhao J, Di Z, et al. Spatially resolved in vivo imaging of inflammation-associated mRNA via enzymatic fluorescence amplification in a molecular beacon[J]. Nature Biomedical Engineering, 2022.(IF 26.8)• Tang S, Guo N, Tang Q, et al. Pyruvate transporter BnaBASS2 impacts seed oil accumulation in Brassica napus[J]. Plant Biotechnology Journal, 2022. (IF 10.1)• Wu K, Wang S, Song W, et al. Enhanced sustainable green revolution yield via nitrogen-responsive chromatin modulation in rice[J]. Science, 2020. (IF 45.8)全式金生物的产品再度亮相 Nature 期刊,不仅是对全式金生物产品卓越品质与雄厚实力的有力见证,更是生动展现了全式金生物长期秉持的“品质高于一切,精品服务客户”核心理念。一直以来,全式金生物凭借对品质的执着追求和对创新的不懈探索,其产品已成为众多科研工作者信赖的得力助手。展望未来,我们将持续推出更多优质产品,期望携手更多科研领域的杰出人才,共同攀登科学高峰,书写科研创新的辉煌篇章。
2025-07-25
文章信息文章题目Remodelling autoactive NLRs for broad-spectrum immunity in plants期刊Nature发表时间2025 年 7 月 16 日主要内容清华大学生命科学学院刘玉乐实验室在 Nature 杂志上发表了题为“Remodelling autoactive NLRs for broad-spectrum immunity in plants”的研究论文,开创性地建立了一种简单高效的人工设计植物抗病基因的全新策略,利用蛋白质工程,将一种携带病原蛋白酶识别切割位点(PCS)的多肽与激活型植物 NLR 免疫受体(aNLR)融合,使其保持失活状态,直到融合蛋白被病原体蛋白酶切割,释放出 aNLR,激活强烈的免疫反应,可使植物获得广谱、持久且完全的抗病。原文链接https://www-nature-com.libproxy1.nus.edu.sg/articles/s41586-025-09252-z使用 TransGen 产品TransZol Up (ET111)TransScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AT311)研究背景植物病原对作物生长和产量构成毁灭性威胁,培育抗病作物是保障全球粮食安全与生态可持续发展的迫切需求。 近年来,重塑植物免疫受体以获得新型抗病基因已成为应对这一威胁的重要策略,然而,现有方法受限于病原的快速进化,往往难以实现广谱持久的抗病。植物 NLR 免疫受体主要包括 TNL(TIR-NLR)、CNL(CC-NLR)和 RNL(CCR-NLR)。其中,TNL 和 CNL 负责识别病原效应因子,RNL 在免疫信号传导中发挥关键作用。近年的研究表明,CNL 和 RNL 在识别病原入侵后可组装形成抗病小体,在膜上形成钙离子通道激活免疫反应,这一过程依赖其 CC 或 CCR 结构域的氨基(N)端序列的完整性。与此一致的是,刘玉乐团队十年前就发现,在 CNL 的 N 端融合额外多肽会抑制其功能。另一方面,在 NLR 的 MHD 基序或其他关键区域引入特定突变,可产生自激活型植物 NLR 免疫受体(autoactive NLR, aNLR)。此外,全世界约 45% 的植物病毒编码其侵染所必需的蛋白酶,大量细菌、真菌、卵菌、线虫和刺吸式昆虫也依赖向植物细胞分泌蛋白酶致病。文章概述研究团队在植物中表达一种 C 端携带病原蛋白酶识别切割位点(PCS)的多肽与激活型植物 NLR 免疫受体(aNLR)的 N 端融合的蛋白。无病原存在时,aNLR 被融合多肽抑制,保持失活状态;当病原入侵时,其编码或分泌的蛋白酶特异切割融合蛋白,释放aNLR,从而激活强烈免疫反应,诱发植物对病原的抗性。若选用保守性高的蛋白酶识别切割位点,该策略可赋予植物广谱且持久的抗病性。研究团队进一步通过实验证明,单个工程化改造的 NLR 受体即可赋予植物(例如模式植物本生烟草、经济作物大豆)对多种马铃薯 Y 病毒属病毒的广谱完全抗性。鉴于不同界的多种病原生物(包括病毒、细菌、卵菌、真菌、线虫和刺吸式昆虫)均编码蛋白酶,这一策略具有望成为植物抗病工程的通用策略,实现对多类病原的抵抗。人工设计抗病基因示意图该策略构建抗病基因具有多重优势:构建简单(仅需改造单个 aNLR 基因)、可针对大量不同的病原定制抗病基因、抗性广谱且持久稳定、不易被病原突破,且抗病效果强(对病毒等病原可实现完全免疫)。此外,该方法具有高度普适性,适用于所有作物,并可与基因组编辑技术结合,直接编辑植物 NLR 基因获得新抗病基因。全式金生物产品支撑优质的试剂是科学研究的利器。全式金生物的高性能 RNA 提取试剂(ET111)及反转录试剂(AT311)助力本研究。产品自上市以来,深受客户青睐,多次荣登知名期刊,助力科学研究。TransZol Up (ET111)本产品利用异硫氰酸胍裂解细胞,与其它总 RNA 提取试剂相比,裂解能力强、速度快,RNA 的提取量与纯度更高。 产品特点 ★ 操作安全性提高:使用 RNA Extraction Agent 替代了氯仿。★ 适用于快速提取多种组织和细胞中的总 RNA。★ 应用范围广:动物、植物组织、血液和细菌等样品。小量样品 (50-100 mg 组织、5×106 细胞、200 μl 血液)。大量样品 (≥1 g 组织或≥107 细胞)。★ 提取速度快:一个小时内可完成反应。★ 操作可视化:溶液呈粉红色,便于分离水相和有机相。★ 提取纯度高:DNA 和蛋白质的污染低。★ RNA 溶解液:便于 RNA 保存和降低对反转录反应的抑制。TransScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AT311)本产品以 RNA 为模板,在同一反应体系中,合成第一链 cDNA 的同时去除 RNA 模板中残留的基因组 DNA。反应结束后,只需在 85℃ 加热 5 秒钟,即可同时失活 TranScript® RT/RI 与 gDNA Remover。 产品特点 ★ 在同一反应体系中,同时完成反转录与基因组 DNA 的去除,操作简便,降低污染机率。★ 产物用于 qPCR:反转录 15 分钟;产物用于 PCR:反转录 30 分钟。★ 反应结束后,同时热失活 RT/RI 与 gDNA Remover。★ 合成片段≤12 kb。产品文献集锦使用 TransZol Up (ET111) 产品发表的部分文章• Wang J Z, Chen T Y, Zhang Z D, et al. Remodelling autoactive NLRs for broad-spectrum immunity in plants [J]. Nature, 2025.(IF 48.5)• Zhou X, Wang G, Tian C, et al. Inhibition of DUSP18 impairs cholesterol biosynthesis and promotes anti-tumor immunity in colorectal cancer[J]. Nature Communications, 2024.(IF 14.7)• Zhou L, Jiang L, Li L, et al. A germline-to-soma signal triggers an age-related decline of mitochondrial stress response[J]. Nature Communications, 2024.(IF 14.7)• Ran R, Li L, Cheng P, et al. High frequency of melanoma in cdkn2b-/-/tp53-/-Xenopus tropicalis[J]. Theranostics, 2024.(IF 13.3)• Duan H J, Chu H Q, Cao T M, et al. Investigation of the cell composition and gene expression in the delayed-type hypersensitivity tuberculin skin test[J]. Military Medical Research, 2023.(IF 34.91)• Liu Y, Chen Y, Zhao Q, et al. A positive TGF‐β/miR‐9 regulatory loop promotes the expansion and activity of tumour‐initiating cells in breast cancer[J]. British Journal of Pharmacology, 2023.(IF 9.47)• Xu C, Ma H, Gao F, et al. Screening of organophosphate flame retardants with placentation-disrupting effects in human trophoblast organoid model and characterization of adverse pregnancy outcomes in mice[J]. Environmental health perspectives, 2022.(IF 9.8)使用 TransScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AT311)产品发表的部分文章• Wang J Z, Chen T Y, Zhang Z D, et al. Remodelling autoactive NLRs for broad-spectrum immunity in plants [J]. Nature, 2025.(IF 48.5)• Zhao P Z, Yang H, Sun J Y, et al. Targeted MYC2 stabilization confers citrus Huanglongbing resistance [J]. Science, 2025.(IF 44.7)• Wang Y, Wang Y, Zhu Y, et al. Angiomotin cleavage promotes leader formation and collective cell migration[J]. Developmental Cell, 2025.(IF 10.7)• Zhao K, Zhang J, Fan Y, et al. PSC1, a basic/helix–loop–helix transcription factor controlling the purplish‐red testa trait in peanut[J]. Journal of Integrative Plant Biology, 2025.(IF 9.3)• Wang S, Du Y, Zhang B, et al. Transplantation of chemically induced pluripotent stem-cell-derived islets under abdominal anterior rectus sheath in a type 1 diabetes patient[J]. Cell, 2024.(IF 45.5)• Zhu J, Zhong X, He H, et al. Generation of human expandable limb-bud-like progenitors via chemically induced dedifferentiation[J]. Cell Stem Cell, 2024. (IF 19.8)• Hong Y, Yu Z, Zhou Q, et al. NAD+ deficiency primes defense metabolism via 1O2-escalated jasmonate biosynthesis in plants[J]. Nature Communications, 2024. (IF 14.7)• Zhang H, Ma J, Wu Z, et al. BacPE: a versatile prime-editing platform in bacteria by inhibiting DNA exonucleases[J]. Nature Communications, 2024.(IF 14.7)• Zuo F, Jiang L, Su N, et al. Imaging the dynamics of messenger RNA with a bright and stable green fluorescent RNA[J]. Nature Chemical Biology, 2024.(IF 12.9)• Gong Q, Wang Y, He L, et al. Molecular basis of methyl-salicylate-mediated plant airborne defence[J]. Nature, 2023.(IF 50.5)• Fan H, Quan S, Ye Q, et al. A molecular framework underlying low-nitrogen-induced early leaf senescence in Arabidopsis thaliana[J]. Molecular Plant, 2023.(IF 17.1)• Wang Y, Wang Z, Chen W, et al. A KDPG sensor RccR governs Pseudomonas aeruginosa carbon metabolism and aminoglycoside antibiotic tolerance[J]. Nucleic Acids Research, 2023.(IF 16.6)• Zhang W, Pan X, Xu Y, et al. Mevalonate improves anti-PD-1/PD-L1 efficacy by stabilizing CD274 mRNA[J]. Acta Pharmaceutica Sinica B, 2023.(IF 14.7)• Liu W, Yao Q, Su X, et al. Molecular insights into Spindlin1-HBx interplay and its impact on HBV transcription from cccDNA minichromosome[J]. Nature Communications, 2023.(IF 14.7)• Yan Z W, Chen F Y, Zhang X, et al. Endocytosis-mediated entry of a caterpillar effector into plants is countered by Jasmonate[J]. Nature Communications, 2023.(IF 14.7)• Guan J, Wang G, Wang J, et al. Chemical reprogramming of human somatic cells to pluripotent stem cells[J]. Nature, 2022.(IF 50.5)• Chen J, Ou Y, Luo R, et al. SAR1B senses leucine levels to regulate mTORC1 signalling[J]. Nature, 2021.(IF 50.5)全式金生物的产品再度亮相 Nature 期刊,不仅是对全式金生物产品卓越品质与雄厚实力的有力见证,更是生动展现了全式金生物长期秉持的“品质高于一切,精品服务客户”核心理念。一直以来,全式金生物凭借对品质的执着追求和对创新的不懈探索,其产品已成为众多科研工作者信赖的得力助手。展望未来,我们将持续推出更多优质产品,期望携手更多科研领域的杰出人才,共同攀登科学高峰,书写科研创新的辉煌篇章。
100 项与 北京全式金生物技术股份有限公司 相关的药物交易
登录后查看更多信息
100 项与 北京全式金生物技术股份有限公司 相关的转化医学
登录后查看更多信息
组织架构
使用我们的机构树数据加速您的研究。
登录
或

管线布局
2025年11月08日管线快照
无数据报导
登录后保持更新
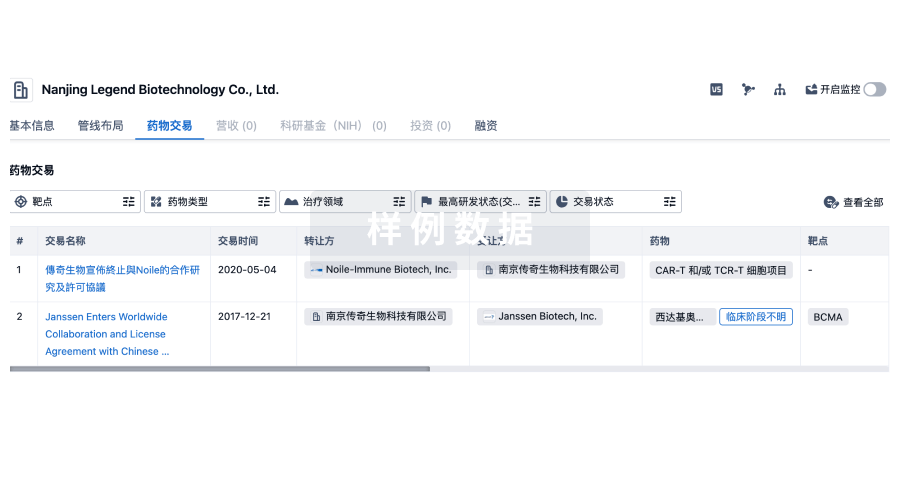
药物交易
使用我们的药物交易数据加速您的研究。
登录
或
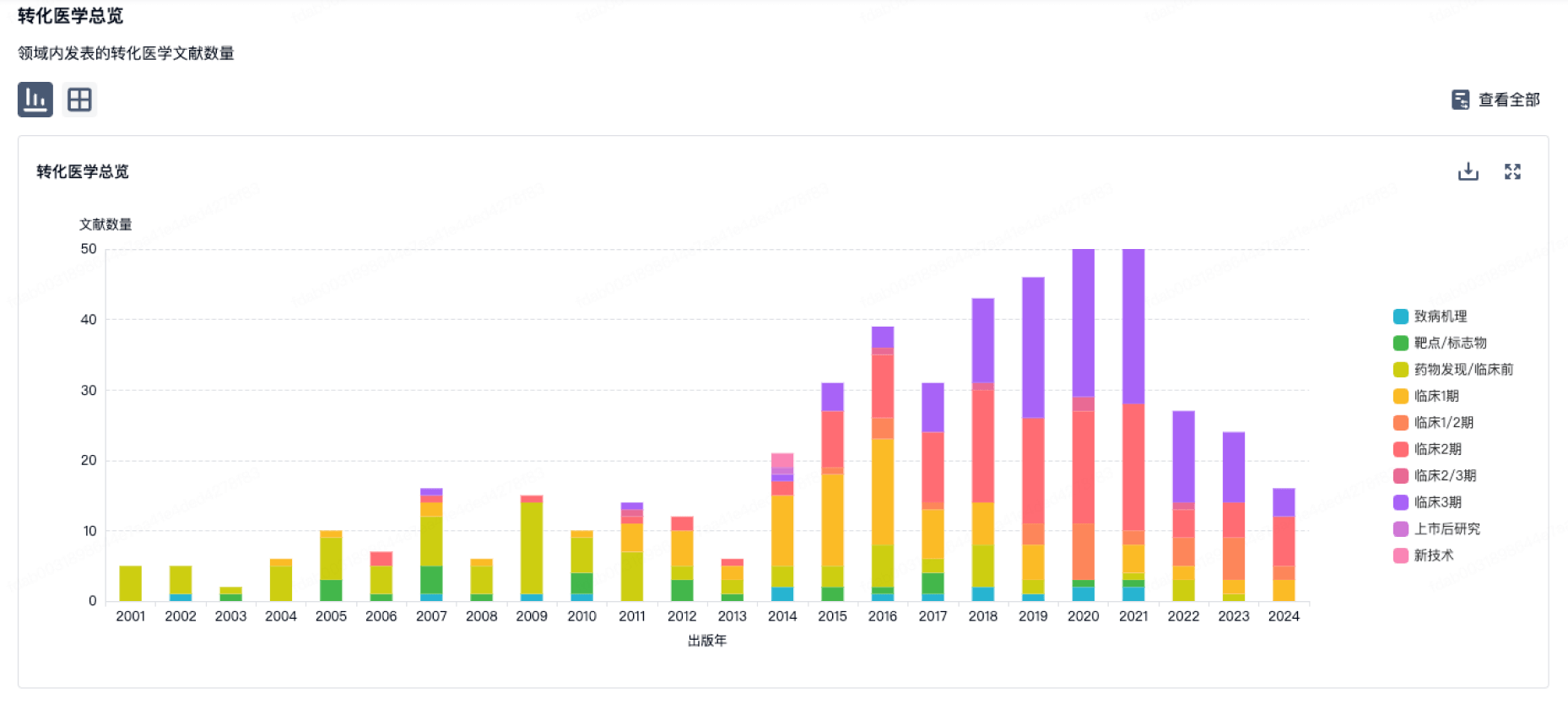
转化医学
使用我们的转化医学数据加速您的研究。
登录
或
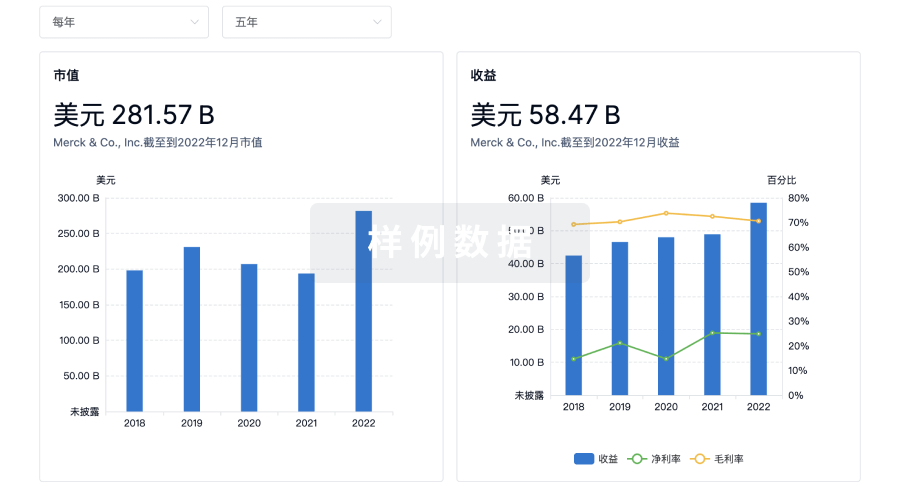
营收
使用 Synapse 探索超过 36 万个组织的财务状况。
登录
或
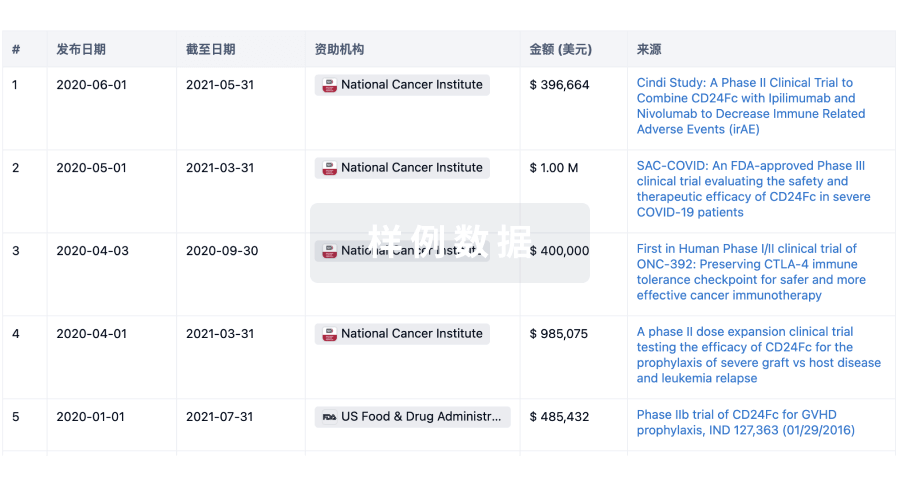
科研基金(NIH)
访问超过 200 万项资助和基金信息,以提升您的研究之旅。
登录
或
投资
深入了解从初创企业到成熟企业的最新公司投资动态。
登录
或
融资
发掘融资趋势以验证和推进您的投资机会。
登录
或
生物医药百科问答
全新生物医药AI Agent 覆盖科研全链路,让突破性发现快人一步
立即开始免费试用!
智慧芽新药情报库是智慧芽专为生命科学人士构建的基于AI的创新药情报平台,助您全方位提升您的研发与决策效率。
立即开始数据试用!
智慧芽新药库数据也通过智慧芽数据服务平台,以API或者数据包形式对外开放,助您更加充分利用智慧芽新药情报信息。
生物序列数据库
生物药研发创新
免费使用
化学结构数据库
小分子化药研发创新
免费使用